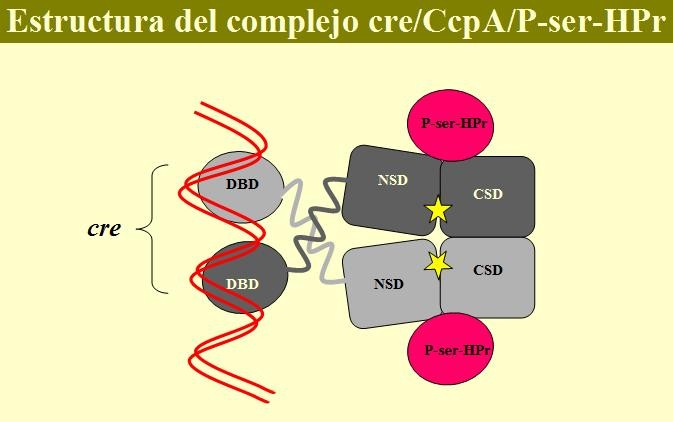
La búsqueda del regulador o reguladores transcripcionales que mediaban la RC en Bacillus subtilis, y que posiblemente actuaban al nivel de la unión a los sitios cre, dio lugar al aislamiento del gen ccpA (catabolite control protein A) (Henkin et al., 1991). ccpA codifica una proteína reguladora de unos 330 aminoácidos perteneciente a la familia de reguladores transcripcionales LacI-GalR. Los reguladores de esta familia presentan un dominio amino-terminal del tipo hélice-vuelta-hélice, implicado en el reconocimiento del DNA, a la vez que otros dominios carboxi-terminales que juegan un papel en la dimerización o tetramerización del regulador, así como la unión de sus moléculas efectoras, que suelen ser del tipo carbohidrato o carbohidrato-fosfato (Weickert & Adhya, 1992). Las a-hélices del dominio de unión al DNA son extremadamente sensibles a mutaciones y cambios en sus aminoácidos suelen producir proteínas CcpA no funcionales (Kuster et al, 1999a). Por el contrario, se han aislado mutantes puntuales en el dominio carboxi-terminal que dan como resultados proteínas CcpA activas en la RC aún en ausencia de glucosa (RC constitutiva) (Kuster et al, 1999b). CcpA ha sido caracterizado en varias bacterias lácticas y relacionadas como L. casei, L. delbrueckii, L. pentosus, L. plantarum, Lactococcus lactis, Staphylococcus xylosus, Enterococcus faecalis y varias especies del género Streptococcus (Egeter & Bruckner, 1996; Stucky et al., 1996; Monedero et al., 1997; Luesink et al., 1998; Leboeuf et al., 2000a; Mahr et al., 2000; van den Bogaard et al., 2000; Muscariello et al., 2001). La organización génica de ccpA en bacterias lácticas es monocistrónica mientras que en el género Bacillus ccpA forma un operón con los genes motAB, dos homologos a genes implicados en la rotación del flagelo, pero de función desconocida. En bacterias lácticas el gen ccpA suele encontrase agrupado junto con un gen que codifica una prolil-aminopeptidasa (pepQ). En L. casei, L. pentosus y L. delbrueckii, Lc. lactis y Streptococcus thermophilus, el orden génico es pepQ-ccpA, con los dos genes transcritos de manera divergente, mientras que en L. plantarum el orden es ccpA–pepQ, con los dos genes transcritos en el mismo sentido. La asociación de ccpA con genes del metabolismo de aminoácidos podría reflejar una unión entre la regulación del metabolismo del carbono y el nitrógeno, como se discutirá más adelante.
CcpA es un regulador transcripcional que precisa de una señal que desencadene su unión a los sitios cre presentes en los genes regulados por catabolito. Esta señal es transmitida por una proteína co-reguladora perteneciente al sistema PTS: la proteína HPr en su forma fosforilada en la serina localizada en la posición 46 de la proteína (Deustcher et al., 1995; Fujita et al., 1995; Jones et al., 1997). P-Ser-HPr interacciona con CcpA activando su unión a los sitios cre, aunque, como se ha podido comprobar en algunos organismos, P-Ser-HPr no es la única molécula señalizadora, y algunos compuestos como FBP, glucosa-6-P ó NADP también pueden influenciar la unión de CcpA a los sitios cre (Gosseringer et al., 1997; Kim et al., 1998). CcpA participa tanto en la RC como en la AC. La unión a cre situados en el promotor o dentro del propio gen o genes regulados reprime la transcripción, seguramente al impedir la unión del complejo de la RNA polimerasa o simplemente bloqueando la elongación del RNA mensajero. Por el contrario, la unión de CcpA a cre situados en posición 5´ respecto al promotor activa la transcrición. Debido a su modo de acción, dado que CcpA activa su unión al DNA únicamente cuando su cofactor esta presente, cabría esperar que se produjese en la célula de una manera constante y uniforme, independientemente de las condiciones de crecimiento. Este parece ser el caso en B. subtilis, donde la síntesis de CcpA es constitutiva. En L. casei y S. thermophilus, el gen ccpA también es transcrito de manera constitutiva, mientras que en L. pentosus, L. plantarum, L. delbrueckii y S. xylosus, la presencia de sitios cre en el propio promotor de ccpA hace que exista una autorepresión en la síntesis de la proteína en presencia de glucosa, lo cual denota un control muy preciso de las cantidades de CcpA presentes en la célula según las condiciones de crecimiento (Egeter & Bruckner, 1996; Mahr et al., 2000; Morel et al., 2001).
Se han obtenido mutantes ccpA por disrupción génica en L. casei, L. pentosus, Lc. lactis, S. thermophilus, S. xylosus y E. faecalis, demostrándose en todos los casos una perdida de la regulación por catabolito de los genes implicados en la utilización de fuentes de carbono secundarias, como b-galactosidasa, b-glucuronidasa, N-acetyl-glucosaminidasa, a-glucosidasa, ó transportadores encargados de internalizar azúcares en la célula ó una pérdida de activación de genes del metabolismo central. Esto demuestra que el modelo de RC/AC mediado por CcpA y establecido para Bacillus parece ser común para el resto de Gram-positivos de bajo contenido en G+C. Sin embargo, el modelo de regulación por catabolito en el que sólo un regulador transcripcional global (CcpA) esté implicado parece resultar una mera simplificación. En B. subtilis se ha descrito la participación de otros reguladores, denominados CcpB y CcpC (Chauvaux et al., 1998; Jourlin-Castelli et al., 2000), que intervienen en el proceso de RC en determinadas circunstancias. Por otra parte, el género Bacillus posee otra proteína homóloga a HPr, llamada Crh, que es a su vez fosforilada en la serina 46 y también participa en la RC que media CcpA (Galinier et al., 1997). Aunque estos reguladores adicionales no se han encontrado en otras bacterias de las que se conoce el genoma completo como L. lactis o L. plantarum.
Referencias:
Chauvaux S., I.T. Paulsen & M.H. Jr Saier. 1998. CcpB, a novel transcription factor implicated in catabolite repression in Bacillus subtilis. J Bacteriol 180:491-497.
Deutscher J., E. Kuster, U. Bergstedt, V. Charrier & W. Hillen. 1995. Protein kinase-dependent HPr/CcpA interaction links glycolytic activity to carbon catabolite repression in gram-positive bacteria. Mol Microbiol 15:1049-1053.
Egeter O. & R. Bruckner. 1996. Catabolite repression mediated by the catabolite control protein CcpA in Staphylococcus xylosus. Mol Microbiol 21:739-749.
Fujita, Y., Y. Miwa, A. Galinier & J. Deutscher. 1995. Specific recognition of the Bacillus subtilis gnt cis-acting catabolite-responsive element by a protein complex formed between CcpA and seryl-phosphorylated HPr. Mol Microbiol 17:953-960.
Galinier, A., J. Haiech, M.C. Kilhoffer, M. Jaquinod, J. Stulke, J. Deutscher & I. Martin-Verstraete. 1997. The Bacillus subtilis crh gene encodes a HPr-like protein involved in carbon catabolite repression. Proc Natl Acad Sci U S A 94:8439-8444.
Gosseringer R., E. Kuster, A. Galinier, J. Deutscher & W. Hillen. 1997. Cooperative and non-cooperative DNA binding modes of catabolite control protein CcpA from Bacillus megaterium result from sensing two different signals. J Mol Biol 266:665-676.
Henkin T.M., F.J. Grundy, W.L. Nicholson & G.H. Chambliss. 1991. Catabolite repression of alpha-amylase gene expression in Bacillus subtilis involves a trans-acting gene product homologous to the Escherichia coli Lacl and GalR repressors. Mol Microbiol 5:575-584.
Jones, B.E., V. Dossonnet, E. Kuster, W. Hillen, J. Deutscher & R.E. Klevit. 1997. Binding of the catabolite repressor protein CcpA to its DNA target is regulated by phosphorylation of its corepressor HPr. J Biol Chem 272:26530-26535.
Jourlin-Castelli, C., N. Mani, M.M. Nakano & A.L. Sonenshein. 2000. CcpC, a novel regulator of the LysR family required for glucose repression of the citB gene in Bacillus subtilis. J Mol Biol 295:865-78.
Kim J.H., M.I. Voskuil & G.H. Chambliss. 1998. NADP, corepressor for the Bacillus catabolite control protein CcpA. Proc Natl Acad Sci U S A 95:9590-9595.
Kuster E., T. Hilbich, M.K. Dahl & W. Hillen. 1999a. Mutations in catabolite control protein CcpA separating growth effects from catabolite repression. J Bacteriol 181:4125-8.
Kuster E., A. Wagner, U. Volker & W. Hillen. 1999b. Mutations in catabolite control protein CcpA showing glucose-independent regulation in Bacillus megaterium. J Bacteriol 181:7634-8.
Leboeuf C., Y. Auffray & A. Hartke. 2000a. Cloning, sequencing and characterization of the ccpA gene from Enterococcus faecalis. Int J Food Microbiol 55:109-113.
Luesink, E.J., R.E. van Herpen, B.P. Grossiord, O.P. Kuipers & W.M de Vos. 1998. Transcriptional activation of the glycolytic las operon and catabolite repression of the gal operon in Lactococcus lactis are mediated by the catabolite control protein CcpA. Mol Microbiol 30:789-798.
Mahr, K., W. Hillen & F. Titgemeyer. 2000. Carbon catabolite repression in Lactobacillus pentosus: analysis of the ccpA region. Appl Environ Microbiol 66:277-283.
Monedero, V., M.J. Gosalbes & G. Pérez-Martínez. 1997. Catabolite repression in Lactobacillus casei ATCC 393 is mediated by CcpA. J Bacteriol 179:6657-6664.
Morel, F., M. Lamarque, I Bissardon, D. Atlan & A. Galinier. 2001. Autoregulation of the biosynthesis of the CcpA-like protein, PepR1, in Lactobacillus delbrueckii subsp bulgaricus. J Mol Microbiol Biotechnol 3:63-66.
Muscariello L., R. Marasco, M. De Felice & M. Sacco.2001.The functional ccpA gene is required for carbon catabolite repression in Lactobacillus plantarum. Appl Environ Microbiol 67:2903-2907.
Stucky K., J. Schick, J.R. Klein, B. Henrich & R. Plapp. 1996. Characterization of pepR1, a gene coding for a potential transcriptional regulator of Lactobacillus delbrueckii subsp. lactis DSM7290. FEMS Microbiol Lett 136:63-69.
van den Bogaard P.T., M. Kleerebezem, O.P. Kuipers & W.M de Vos. 2000. Control of lactose transport, beta-galactosidase activity, and glycolysis by CcpA in Streptococcus thermophilus: evidence for carbon catabolite repression by a non-phosphoenolpyruvate-dependent phosphotransferase system sugar. J Bacteriol 182:5982-5989.
Weickert M.J. & S. Adhya. 1992. A family of bacterial regulators homologous to Gal and Lac repressors. J Biol Chem 267:15869-15874.